在 Intel Gaudi 2 上加速蛋白质语言模型 ProtST

简介
蛋白质语言模型 (PLM) 已成为预测和设计蛋白质结构和功能的强大工具。在 2023 年国际机器学习大会 (ICML) 上,MILA 和 Intel Labs 发布了 ProtST,这是一种基于文本提示的开创性多模态蛋白质设计语言模型。自那时起,ProtST 在研究界广受欢迎,在不到一年的时间里被引用超过 40 次,显示了该工作的科学实力。
PLM 最流行的任务之一是预测氨基酸序列的亚细胞定位。在此任务中,用户将氨基酸序列输入模型,模型输出一个标签,指示该序列的亚细胞定位。开箱即用的零样本 ProtST-ESM-1b 优于最先进的少样本分类器。
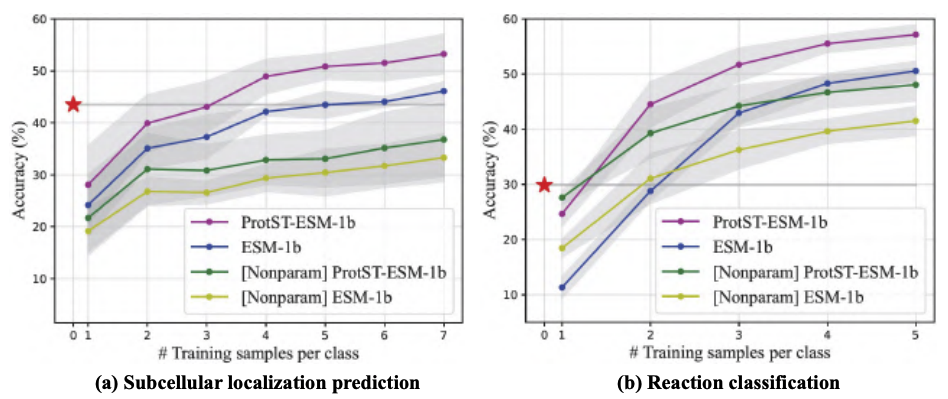
为了使 ProtST 更易于访问,Intel 和 MILA 重新架构并在 Hugging Face Hub 上共享了该模型。您可以在此处下载模型和数据集。
本文将向您展示如何使用 Intel Gaudi 2 加速器和 Optimum for Intel Gaudi 开源库高效运行 ProtST 推理并对其进行微调。Intel Gaudi 2 是英特尔设计的第二代 AI 加速器。请查看我们的上一篇博客文章,了解深入介绍以及通过 Intel Developer Cloud 访问它的指南。借助 Optimum for Intel Gaudi 库,您只需进行最少的代码更改即可将基于 transformer 的脚本移植到 Gaudi 2。
ProtST 推理
常见的亚细胞定位包括细胞核、细胞膜、细胞质、线粒体等,此数据集中对此有更详细的描述。
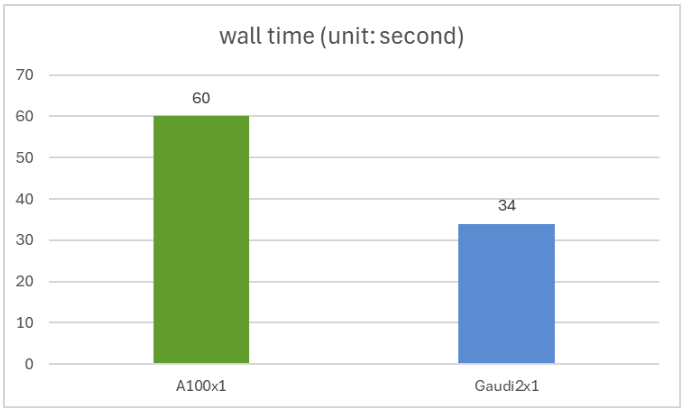
我们使用 ProtST-SubcellularLocalization 数据集的测试集,比较 ProtST 在 NVIDIA A100 80GB PCIe 和 Gaudi 2 加速器上的推理性能。该测试集包含 2772 个氨基酸序列,序列长度从 79 到 1999 不等。
您可以使用此脚本重现我们的实验,其中我们以全 bfloat16 精度运行模型,批处理大小为 1。我们在 Nvidia A100 和 Intel Gaudi 2 上获得了相同的 0.44 精度,Gaudi2 的推理速度比 A100 快 1.76 倍。单个 A100 和单个 Gaudi 2 的实际时间如下图所示。
ProtST 微调
在下游任务上对 ProtST 模型进行微调是提高建模准确性的一种简单且成熟的方法。在此实验中,我们将模型专门用于二元定位,这是亚细胞定位的一个更简单的版本,其二元标签指示蛋白质是膜结合还是可溶的。
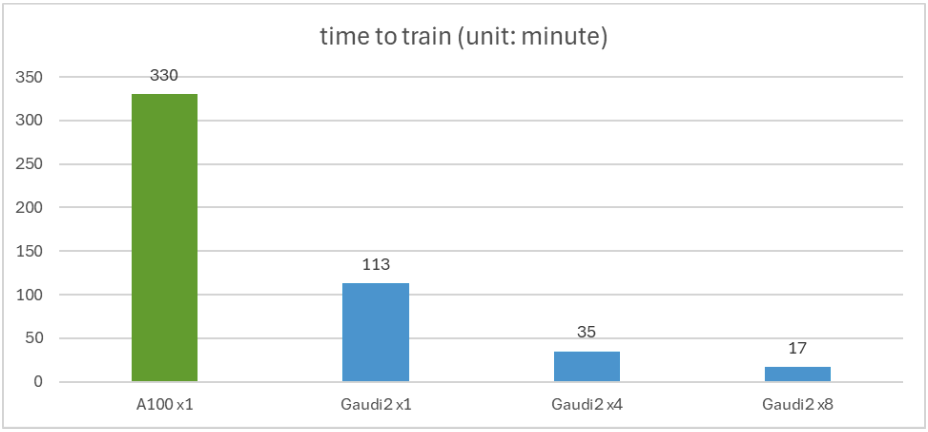
您可以使用此脚本重现我们的实验。在这里,我们以 bfloat16 精度在 ProtST-BinaryLocalization 数据集上对 ProtST-ESM1b-for-sequential-classification 模型进行微调。下表显示了在不同训练硬件设置下测试集上的模型准确性,它们与论文中发布的 MLLM 结果(约 92.5% 的准确性)非常接近。

下图显示了微调时间。单个 Gaudi 2 比单个 A100 快 2.92 倍。该图还显示了分布式训练如何通过 4 或 8 个 Gaudi 2 加速器近乎线性地扩展。
结论
在这篇博客文章中,我们展示了在基于 Optimum for Intel Gaudi 加速器的 Gaudi 2 上部署 ProtST 推理和微调的简易性。此外,我们的结果显示了与 A100 相比具有竞争力的性能,推理速度提高了 1.76 倍,微调速度提高了 2.92 倍。以下资源将帮助您开始在 Intel Gaudi 2 加速器上使用您的模型:
感谢您的阅读!我们期待看到您基于 Intel Gaudi 2 加速器功能,在 ProtST 之上构建的创新成果。

