从Protein LLaMA开始小规模尝试
上个月我发布了一个蛋白质嵌入模型。通过一个新的植物蛋白质数据集(GreenBeing),我正在探索训练一组结合蛋白质和自然语言文本的模型。生物信息学研究通常关注蛋白质的功能、在细胞内的位置以及在生物体内的位置。与更大的自然语言模型结合可能会实现问答、聊天、新蛋白质的描述和解释。
tinyllama-mixpretrain-quinoa-sciphi
这是一个1.1B的TinyLLaMA模型,我使用Trainer在数千种藜麦蛋白质和科学文本片段(SciPhi/textbooks-are-all-you-need-lite)的混合数据上继续训练。虽然我曾考虑添加“生物令牌”来专门表示氨基酸,但我尚未尝试。我分享了训练用的CoLab笔记本。
今天我尝试使用PEFT对这个模型进行微调,数据来自GreenBeing微调分割中其他植物的蛋白质。我希望将来能将它DSPy化/做成问答格式,但目前是这样:
<MMNPDGGDGDR…> 功能\n位置\n其他注释
我排除了来自玉米属(Zea genus)的蛋白质,以便评估模型。笔记本链接请看这里,最终LoRA请看这里。
如果这个项目能取得有意义的结果,我将尝试使用更大的模型和数据集(LLaMA 3或Mixtral,GreenBeing的完整预训练分割)进行训练。我还不确定PEFT的新令牌功能是否足以支持生物令牌。此外,有一段时间我尝试过将MergeKit用于仅含蛋白质的TinyLLaMA和斯坦福大学的BioMedLM,或PharMolix/BioMedGPT-LM-7B的16位版本。它们的架构和参数不匹配。如果对此有任何建议,请告诉我。🙏
GreenBeing数据集详情
GreenBeing是一个包含食物作物及其野生近缘种蛋白质的数据集。我决定这样组织这个数据集:
- 预训练分割:来自UniProt上选定作物物种及其野生近缘种的氨基酸序列
- 微调分割:来自相同分类群的已审查序列,以及UniProtKB(知识库)中的文本注释
- 研究分割:99%为藜麦,其余为小藜和苋菜
理解UniProt 数据
你在杂货店找到的大部分大米是普通水稻(Oryza sativa)。其粳稻亚种在已审阅的SwissProt数据集中有约4,100个蛋白质,在未审阅的TrEMBL中有约44,700个。这比Google和UniProt所说的水稻基因数量还要多,所以我不知道这是否是重复、变体还是错误。从阅读“未审阅”的含义中,我确信只要我们不使用任何自动化/预测的注释,就可以在预训练中使用这些序列。
这些序列使用IUPAC-IUB代码,其中字母A-Z对应氨基酸。这与使用核苷酸(ACTG)的基因组数据不同,后者模型可以涵盖基因表达。
注意:在搜索UniProt时,请使用taxonomy_name:__,再加上taxonomy_name:Viridiplantae,这样你就能看到玉米属(Zea)、小麦族(Triticeae)等,而不是包含它们的病毒和病虫害的全文搜索!

预训练分割
我向上追溯水稻的分类树到稻族(Oryzeae),以包含野稻,并且我找到了其他流行作物(玉米、大豆、小麦、番茄)的满意级别。数据集中有一个物种列用于过滤,但是关于用野生近缘种的基因来强化和多样化作物,有有趣的研究。
以下是这组蛋白质的分布,其中蝶形花亚科(Papilionoideae,包括大豆、豌豆、鹰嘴豆、花生等)位居首位
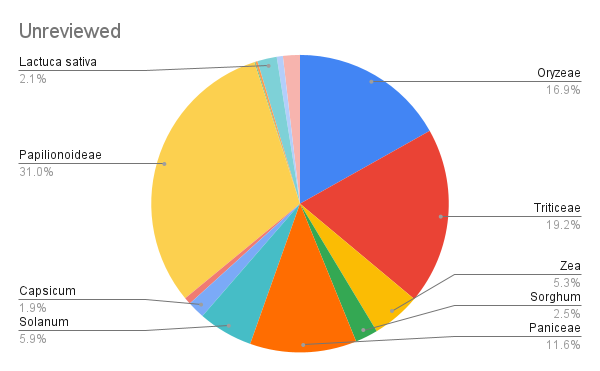
微调分割
我使用相同的分类群来查询已审阅的/SwissProt蛋白质。与其它植物相比,水稻在该数据集中有极好的覆盖率——几乎占微调分割的一半。

研究分割
UniProt有一个接近完整的藜麦蛋白质组,但其状态未经审查——甚至那些让你获得红/白/黑种子的基因仍在鉴定中。你可以使用蛋白质嵌入来将这些蛋白质与其它物种中最近的已审查蛋白质进行匹配。我希望这能为藜麦研究做出贡献,因为它在许多地区被认为是耐旱作物。
限制和安全注意事项
UniProt蛋白质于2024年3月29日下载。
来自相似物种和登录号的已审查和未审查蛋白质之间可能存在显著重叠。
物种包括不可食用的野生近缘种。
许多人对小麦/麸质、茄科植物、玉米及其他作物有过敏反应。
辣椒可能很辣。🌶️